一種基于劃窗映射的基因組測序數據快速注釋方法和系統與流程
1.本發明屬于生物信息技術領域,具體地,涉及一種基于劃窗映射的基因組測序數據快速注釋方法和系統。
背景技術:
2.下一代測序(next-generation sequencing,ngs)又稱為高通量測序(high-throughput sequencing),是基于pcr和基因芯片發展而來的邊合成邊測序技術。高通量測序技術的特點主要有:測序讀長短,通量高,準確度高。高通量測序相比一代測序大幅降低了成本,同時保持了較高準確性,并且大幅降低了測序時間,目前高通量測序已經在全組學得到廣泛應用。比如:有參轉錄組測序,重測序,dna甲基化測序,m6a甲基化測序,單細胞測序等。
3.dna甲基化是表觀遺傳修飾的主要方式,能在不改變dna序列的前提下,改變遺傳表現,在調控基因表達和染質構象等方面發揮著重要作用。dna甲基化主要形成5-甲基胞嘧啶(5-mc)和少量的n6-甲基嘌呤(n6-ma)及7-甲基鳥嘌呤(7-mg)等。通常地,甲基化dna主要指5-甲基胞嘧啶(5mc)。哺乳動物細胞中甲基化主要發生在cg二核苷酸的胞嘧啶上,植物細胞中則存在很大比例的non-cg(chh、chg,h代表a、c、t)甲基化。5-甲基胞嘧啶(5-mc)由dna甲基轉移酶(dna methyl-transferase,dnmt)催化s-腺苷甲硫氨酸(s-adenosylmethionine,sam)作為甲基供體,將胞嘧啶轉變為5-甲基胞嘧啶(mc)。
4.全基因組甲基化測序(whole-genome bisulphite sequencing,wgbs)結合亞硫酸氫鹽轉化(bisulfite conversion)方法與二代測序技術,可在單堿基分辨率水平上高效地檢測全基因組dna甲基化狀態。亞硫酸氫鹽處理可以使dna中未發生甲基化的胞嘧啶脫氨基轉變成尿嘧啶,而甲基化的胞嘧啶保持不變;pcr擴增所需片段,則尿嘧啶全部轉化成胸腺嘧啶。對pcr產物進行高通量測序,與參考序列比對,即可判斷cpg/chg/chh位點是否發生甲基化。全基因組甲基化測序可全面、精確地檢測全基因組dna甲基化狀態,為更深入的表觀遺傳調控分析奠定基礎。
5.基因啟動子區cpg island通常為去甲基化狀態,促進基因轉錄,而異常甲基化會導致轉錄的失活。一般而言,cpg island甲基化會導致基因沉默。dna甲基化在基因組印記方面有重要作用,在雙等位基因中的一個發生高甲基化會導致單等位基因表達。
6.目前的生物信息軟件對于dna甲基化測序數據在啟動子區(promoter),外顯子區(exonic),內含子區(intronic)和基因間區(intergenic)等基因結構區域的注釋和cpg island區域的注釋沒有一致和快速的注釋方法。
技術實現要素:
7.為了解決上述技術問題中的至少一個,本發明采取的技術方案如下:
8.本發明第一方面提供一種基于劃窗映射的基因組測序數據快速注釋方法,包括以下步驟:
9.s1,建立索引文件:
10.獲得測序樣本來源物種的功能組件區的起始位點和終止位點,對于每一個功能組件,利用公式(1)獲得映射值:
[0011][0012]
其中,gi代表第i個位點的映射值,int表示取整數,si代表第i個位點數值,si代表第i個位點所屬的劃窗區間的起始位點,w代表劃窗大小,w大小根據所述來源物種的所有功能組件區長度確定,
[0013]
由此獲得所有功能組件區的起始位點和終止位點的映射值以及位于功能組件區內的窗口起點/終點的映射值,按下列格式構建索引文件:
[0014]
chr s e s e function
[0015]
其中,chr代表功能組件區所在染體位置信息,s代表功能組件區起始位點或者位于功能組件區內的劃窗起點的映射值,e代表功能組件區終止位點或者位于功能組件區內的劃窗終點的映射值,s代表功能組件區起始位點,e代表功能組件區終止位點,function代表功能組件區的類別;
[0016]
s2,獲得待注釋位點的映射值:其位點數值為q,同樣利用公式(1)獲得待注釋位點的映射值g;
[0017]
s3,將步驟s2獲得的映射值g在所述索引文件的第2列和第3列進行搜索,若對于某一行j,g滿足sj≤g≤ej,進一步判斷q是否滿足sj≤q≤ej,若滿足,則所述待注釋位點可注釋位于第j行對應的功能組件區。
[0018]
在本發明的一些實施方案中,所述w的確定方法具體如下:
[0019]
(1)獲得所述來源物種的所有功能組件區的長度;
[0020]
(2)獲得所有功能組件區長度的代表值,根據代表值確定w值。
[0021]
在本發明的一些具體實施方案中,所述代表值選自中位數、眾數、平均數、1/4分位數和3/4分位數中的一種,所述w值代表值。在本發明的另一些具體實施方案中,所述w值位于所述1/4分位數和3/4分位數之間。
[0022]
在這里,w值的獲得是本發明意外發現可以使得處理后注釋效率更高的選取方法,本領域技術人員也可以利用其他方式選取w值,只要不違背本發明的核心思想,都應視為落入本發明的保護范圍。
[0023]
在本發明的一些實施方案中,所述來源物種為哺乳動物。優選地,所述來源物種為人。
[0024]
在本發明的一些實施方案中,所述基因測序數據是指dna甲基化測序數據。
[0025]
在本發明的一些實施方案中,所述功能組件區包括啟動子區、外顯子區、內含子區、promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis、重復區和mirna區。
[0026]
其中,promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis是根據cgi所屬的基因位置進行定義的:
[0027]
promoter cgis-1000bp tss to+300bp tssintragenic cgis+300bp tss to+300bp tes
3'transcript cgis-300bp tes to+300bp tesintergenic cgis-300bp tes to-1000bp next gene's promoter
[0028]
本發明第二方面提供一種基于劃窗映射的基因組測序數據快速注釋系統,包括以下模塊:
[0029]
索引庫模塊,用于存儲索引文件,其中,所述索引文件的構建方法如下:
[0030]
獲得測序樣本來源物種的功能組件區的起始位點和終止位點,對于每一個功能組件,利用公式(1)獲得映射值:
[0031][0032]
其中,gi代表第i個位點的映射值,int表示取整數,si代表第i個位點數值,si代表第i個位點所屬的劃窗區間的起始位點,w代表劃窗大小,w大小根據所述來源物種的所有功能組件區長度確定,
[0033]
由此獲得所有功能組件區的起始位點和終止位點的映射值以及位于功能組件區內的窗口起點/終點的映射值,按下列格式構建索引文件:
[0034]
chr s e s e function
[0035]
其中,chr代表功能組件區所在染體位置信息,s代表功能組件區起始位點或者位于功能組件區內的劃窗起點的映射值,e代表功能組件區終止位點或者位于功能組件區內的劃窗終點的映射值,s代表功能組件區起始位點,e代表功能組件區終止位點,function代表功能組件區的類別,
[0036]
輸入模塊,用于接收測序數據,獲得待注釋位點,并利用公式(1)計算待注釋位點的索引值,
[0037]
搜索模塊,分別與輸入模塊和索引庫模塊連接,用于將輸入模塊獲得的所述待注釋位點的索引值在所述索引文件的第2列和第3列進行搜索,若對于某一行j,g滿足sj≤g≤ej,進一步判斷q是否滿足sj≤q≤ej,若滿足,則所述待注釋位點可注釋位于第j行對應的功能組件區,
[0038]
結果輸出模塊,用于將注釋結果輸出。
[0039]
在本發明的一些實施方案中,所述w的確定方法具體如下:
[0040]
(1)獲得所述來源物種的所有功能組件區的長度;
[0041]
(2)獲得所有功能組件區長度的代表值,根據代表值確定w值。
[0042]
在本發明的一些具體實施方案中,所述代表值選自中位數、眾數、平均數、1/4分位數和3/4分位數中的一種,所述w值代表值。在本發明的另一些具體實施方案中,所述w值位于所述1/4分位數和3/4分位數之間。
[0043]
在本發明的一些實施方案中,所述來源物種為哺乳動物。優選地,所述來源物種為人。
[0044]
在本發明的一些實施方案中,所述基因測序數據是指dna甲基化測序數據。
[0045]
在本發明的一些實施方案中,所述功能組件區包括啟動子區、外顯子區、內含子區、promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis、重復區和mirna區。
[0046]
其中,promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis
是根據cgi所屬的基因位置進行定義的:
[0047]
promoter cgis-1000bp tss to+300bp tssintragenic cgis+300bp tss to+300bp tes3'transcript cgis-300bp tes to+300bp tesintergenic cgis-300bp tes to-1000bp next gene's promoter
[0048]
本發明的有益效果
[0049]
相對于現有技術,本發明具有如下有益效果:
[0050]
利用本發明的方法和系統,通過對功能組件位置進行映射,方法簡單易操作,并可以大幅提高搜索注釋效率。以人類1號染體為例,搜索效率可以提升497506倍。對于多個染體及多個樣本的注釋,效果更加明顯。
附圖說明
[0051]
圖1示出了cgi所屬的基因位置。
[0052]
圖2示出了一個位點(10540)位于一個啟動子區([10300,13000])中的示意圖。
[0053]
圖3示出了本發明基于劃窗映射的基因組測序數據快速注釋系統示意圖。。
具體實施方式
[0054]
除非另有說明、從上下文暗示或屬于現有技術的慣例,否則本技術中所有的份數和百分比都基于重量,且所用的測試和表征方法都是與本技術的提交日期同步的。在適用的情況下,本技術中涉及的任何專利、專利申請或公開的內容全部結合于此作為參考,且其等價的同族專利也引入作為參考,特別這些文獻所披露的關于本領域中的合成技術、產物和加工設計、聚合物、共聚單體、引發劑或催化劑等的定義。如果現有技術中披露的具體術語的定義與本技術中提供的任何定義不一致,則以本技術中提供的術語定義為準。
[0055]
本技術中的數字范圍是近似值,因此除非另有說明,否則其可包括范圍以外的數值。數值范圍包括以1個單位增加的從下限值到上限值的所有數值,條件是在任意較低值與任意較高值之間存在至少2個單位的間隔。例如,如果記載的是100至1000,意味著明確列舉了所有的單個數值,例如100,101,102等,以及所有的子范圍,例如100到166,155到170,198到200等。對于包含小于1的數值或者包含大于1的分數(例如1.1,1.5等)的范圍,則適當地將1個單位看作0.0001,0.001,0.01或者0.1。對于包含小于10(例如1到5)的個位數的范圍,通常將1個單位看作0.1。這些僅僅是想要表達的內容的具體示例,并且所列舉的最低值與最高值之間的數值的所有可能的組合都被認為清楚記載在本技術中。
[0056]
術語“包含”,“包括”,“具有”以及它們的派生詞不排除任何其它的組分、步驟或過程的存在,且與這些其它的組分、步驟或過程是否在本技術中披露無關。為消除任何疑問,除非明確說明,否則本技術中所有使用術語“包含”,“包括”,或“具有”的組合物可以包含任何附加的添加劑、輔料或化合物。相反,出來對操作性能所必要的那些,術語“基本上由
……
組成”將任何其他組分、步驟或過程排除在任何該術語下文敘述的范圍之外。術語“由
……
組成”不包括未具體描述或列出的任何組分、步驟或過程。除非明確說明,否則術語“或”指列出的單獨成員或其任何組合。
[0057]
為了使本發明所解決的技術問題、技術方案及有益效果更加清楚明白,以下結合
實施例,對本發明進行進一步詳細說明。
[0058]
實施例
[0059]
以下例子在此用于示范本發明的優選實施方案。本領域內的技術人員會明白,下述例子中披露的技術代表發明人發現的可以用于實施本發明的技術,因此可以視為實施本發明的優選方案。但是本領域內的技術人員根據本說明書應該明白,這里所公開的特定實施例可以做很多修改,仍然能得到相同的或者類似的結果,而非背離本發明的精神或范圍。
[0060]
除非另有定義,所有在此使用的技術和科學的術語,和本發明所屬領域內的技術人員所通常理解的意思相同,在此公開引用及他們引用的材料都將以引用的方式被并入。
[0061]
那些本領域內的技術人員將意識到或者通過常規試驗就能了解許多這里所描述的發明的特定實施方案的許多等同技術。這些等同將被包含在權利要求書中。
[0062]
下述實施例中的實驗方法,如無特殊說明,均為常規方法。下述實施例中所用的儀器設備,如無特殊說明,均為實驗室常規儀器設備;下述實施例中所用的試驗材料,如無特殊說明,均為自常規生化試劑商店購買得到的。
[0063]
實施例1基于dna甲基化測序進行功能組件的快速注釋方法
[0064]
1.基因組功能組件分類與定義
[0065]
啟動子是一個dna序列,蛋白質與該序列結合以從啟動子下游的dna啟動單個rna轉錄物的轉錄。rna轉錄物可以編碼一種蛋白質(mrna),或者可以具有自身的功能(如trna或rrna)。啟動子位于基因轉錄起始位點附近,位于dna上游(朝向有義鏈的5'區域)。啟動子長度約為100-1000個堿基對,其序列高度依賴于基因和轉錄產物、類型或類別招募到該地點的rna聚合酶和生物種類。啟動子區是rn a聚合酶的結合區,其結構直接關系到轉錄的效率。
[0066]
轉錄起始位點(tss)是指與新生rna鏈第一個核苷酸相對應dna鏈上的堿基,通常為一個嘌呤。常把起點前面,即5'末端的序列稱為上游(upstream),而把其后面即3'末端的序列稱為下游(downstream)。在描述堿基的位置時,一般用數字表示,tss起點為+1,下游方向依次為+2、+3
……
,上游方向依次為-1、-2、-3
……
。
[0067]
在本實施例中,發明人對于啟動子區基于tss進行了統一定義,作為后續的dn a甲基化測序分析的統一注釋標準,如表1所示:
[0068]
表1啟動子區定義
[0069]
啟動子區promoters-2,200至+500bp近端proximal(p)-200至+500bp中端intermediate(i)-200至-1,000bp遠端distal(d)-1,000至-2,200bp
[0070]
啟動子序列中的相對位置和cpg含量是啟動子甲基化程度的重要影響因素,即o/e比值(observed-to-expected cpg ratio),根據該比值發明人將啟動子區cpg的o/e值分為低、中、高(lcp,icp和hcp)三類。計算公式如下:
[0071][0072]
其中,num of cpg表示cpg數量,num of c表示序列中堿基c數量,num of g表示序列中堿基g數量,total number of nucleotides in the sequence表達序列中總堿基數
量。
[0073]
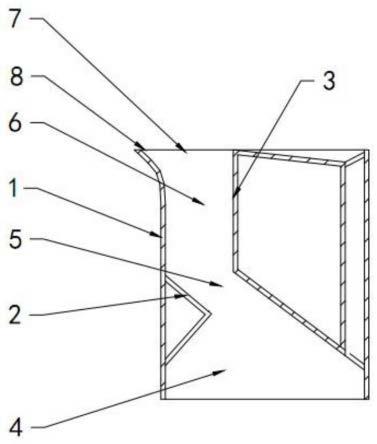
發明人根據cpg島(cgi)所屬的基因位置對cgi進行如表2所述定義:
[0074]
表2cgi定義
[0075][0076][0077]
cgi所屬的基因位置定義如圖1所示。
[0078]
2.利用位點平移法對序列所屬功能組件區進行注釋
[0079]
發明人根據dna甲基化測序的結果,將測序reads比對到基因組上,通常比對結果是以sam格式輸出。sam格式中包括位點位置信息,pos:比對上的最左邊的定位,即reads比對到基因組上的第一個堿基的位置情況。根據這個比對的位置情況,本領域人員需要快速注釋到這個位點是處于基因的哪個功能區間(如啟動子區、外顯子區、內含子區),同時還需要確認是否處于cgi/cgi shores區域。
[0080]
在此,以人的基因組為例,人的1號染體的長度為249250621個堿基,假設某幾條reads比對到該染體的第10540個堿基位置上,如何快速地通過位點搜索到該位置(10540)是否屬于啟動子區成為本領域的一大難題。
[0081]
假設該位點是落在其中的一個基因的啟動子區([10300,13000])內(如圖2所示),如果按照單個位點平移的方法來進行遍歷搜索注釋,那么將循環10540次(從第1個堿基開始遍歷)才能到這個位點位于基因組的啟動子區,該方法執行起來效率非常低。
[0082]
3.劃窗映射法對序列所屬功能組件區進行注釋
[0083]
為了提高功能注釋的搜索效率,發明人創建了一種快速注釋的劃窗映射搜索方法,詳細過程如下:
[0084]
(1)首先,發明人采用如下公式來進行基因組的功能組件的映射創建:
[0085][0086]
其中,gi代表第i個位點的映射值,int表示取整數,si代表第i個位點數值,si代表第i個位點所屬的劃窗(window slide)區間的起始位點,w代表劃窗大小。
[0087]
其中,w的大小根據來源物種的所有功能元件區的長度來確定。具體地:
[0088]
(1)獲得來源物種的所有功能組件區的長度;
[0089]
(2)獲得所有功能組件區長度的代表值,根據代表值確定w值。
[0090]
以人為例,代表值為1/4分位數和3/4分位數,分別是150和2000。在1/4分位數和3/4分位數之間選取w值,經過選取,w=500。
[0091]
以上面的啟動子區[10300,13000]為例,該啟動子區起始位點(10300),si=10300,由劃窗法可知,si=10000,gi=10300/500=20。
[0092]
采用劃窗法可知,該啟動子區位于第一個劃窗的終止位點是10500(10000+500),相應的索引值為:gj=10500/500=21。以此類推,可得到該啟動子區內其他劃窗起點/終點的映射值分別為:11000/500=22、11500/500=23、12000/500=24、12500/50=25、13000/
500=26,該啟動子的終止位點剛好也是一個劃窗的終止位點。
[0093]
發明人以該方法對該啟動子區創建如下的索引進行存儲,格式如下:
[0094]
chrsesefunctionchr120211030013000promoterchr121221030013000promoterchr122231030013000promoterchr123241030013000promoterchr125261030013000promoter
[0095]
其中,chr代表功能組件區所在染體位置信息,s代表功能組件區起始位點或者位于功能組件區內的劃窗起點的映射值,e代表功能組件區終止位點或者位于功能組件區內的劃窗終點的映射值,s代表功能組件區起始位點,e代表功能組件區終止位點,function代表功能組件區的類別。功能組件區的類別包括但不限于:啟動子區(promoter)、外顯子區(exonic)、內含子區(intronic)、promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis、重復區(repeat region)和mirna區。
[0096]
對已知的所有功能組件均進行類似的索引轉換得到索引文件。
[0097]
通過該方法,發明人對于原來的位點進行了數據壓縮,[20,26]該區間的對應的原始位點為:[10000,13000],即用7個位點存儲了原來3000個位點,搜索效率可以提升429倍,對于數據寵大的基因組數據,效果十分明顯。
[0098]
注釋時,只需要進行注釋的位點,也進行相應索引值的轉化后即可進行注釋:
[0099]gk
=k/500
[0100]
其中,gk代表位點k的映射值,sk代表位點k的位點數值。
[0101]
搜索的時候,根據gk到索引文件里去搜索第二列(s)和第二列(e)數據,同時,發明人根據查詢的位點查是否落在第三列和第四列區間內,如果s≤gk≤e,并且s≤sk≤e那么位點k可以注釋成相應的功能組件。
[0102]
例如對于上述10540這個位點,利用上述方法進行相應索引值的轉化,轉化后的數值如下:
[0103]gk
=10540/500=21
[0104]
該位點滿足20≤21≤21,并且10300≤10540≤13000,因此位點k被注釋為啟動子。
[0105]
實施例2基于dna甲基化測序進行功能組件的快速注釋方法的應用
[0106]
本實施例以1號染體為例,1號染體的序列總共有249250621個堿基,如果按傳統的位點遍歷的方法,需要最多遍歷249250621次才能到注釋的位點。而按照實施例1的劃窗映射搜索法,w=500,最多將分為498502個索引值。搜索時最多501次搜索即可以到該位點,搜索效率提升了497506倍。
[0107]
具體的效率提升比較如表3:
[0108]
表3效率比較
[0109]
[0110]
由此可見,利用實施例1的劃窗映射搜索法,可以大幅提升搜索性能。
[0111]
實施例3基于劃窗映射的基因組測序數據快速注釋系統
[0112]
如圖3所示,本實施例提供一種系統,來實現上述快速注釋方法,該系統包括:
[0113]
索引庫模塊,用于存儲上述構建的索引文件
[0114]
輸入模塊,用于接收測序數據,獲得待注釋位點q,并利用上述公式計算待注釋位點q的索引值g,
[0115]
搜索模塊,分別與輸入模塊和索引庫模塊連接,用于將輸入模塊獲得的所述待注釋位點的索引值在所述索引文件的第2列和第3列進行搜索,若對于某一行j,g滿足sj≤g≤ej,進一步判斷q是否滿足sj≤q≤ej,若滿足,則所述待注釋位點可注釋位于第j行對應的功能組件區,
[0116]
結果輸出模塊,用于將注釋結果輸出。
[0117]
在本發明提及的所有文獻都在本技術中引用作為參考,就如同每一篇文獻被單獨引用作為參考那樣。此外應理解,在閱讀了本發明的上述講授內容之后,本領域技術人員可以對本發明作各種改動或修改,這些等價形式同樣落于本技術所附權利要求書所限定的范圍。
技術特征:
1.一種基于劃窗映射的基因組測序數據快速注釋方法,其特征在于,包括以下步驟:s1,建立索引文件:獲得測序樣本來源物種的功能組件區的起始位點和終止位點,對于每一個功能組件,利用公式(1)獲得映射值:其中,g
i
代表第i個位點的映射值,int表示取整數,s
i
代表第i個位點數值,s
i
代表第i個位點所屬的劃窗區間的起始位點,w代表劃窗大小,w大小根據所述來源物種的所有功能組件區長度確定,由此獲得所有功能組件區的起始位點和終止位點的映射值以及位于功能組件區內的窗口起點/終點的映射值,按下列格式構建索引文件:chr s e s e function其中,chr代表功能組件區所在染體位置信息,s代表功能組件區起始位點或者位于功能組件區內的劃窗起點的映射值,e代表功能組件區終止位點或者位于功能組件區內的劃窗終點的映射值,s代表功能組件區起始位點,e代表功能組件區終止位點,function代表功能組件區的類別;s2,獲得待注釋位點的映射值:其位點數值為q,同樣利用公式(1)獲得待注釋位點的映射值g;s3,將步驟s2獲得的映射值g在所述索引文件的第2列和第3列進行搜索,若對于某一行j,g滿足s
j
≤g≤e
j
,進一步判斷q是否滿足s
j
≤q≤e
j
,若滿足,則所述待注釋位點可注釋位于第j行對應的功能組件區。2.根據權利要求1所述的基因組測序數據快速注釋方法,其特征在于,所述w的確定方法具體如下:(1)獲得所述來源物種的所有功能組件區的長度;(2)獲得所有功能組件區長度的代表值,根據代表值確定w值。3.根據權利要求2所述的基因組測序數據快速注釋方法,其特征在于,所述代表值選自中位數、眾數、平均數、1/4分位數和3/4分位數中的一種或者所述w值位于所述1/4分位數和3/4分位數之間。4.根據權利要求1所述的基因組測序數據快速注釋方法,其特征在于,所述來源物種為哺乳動物。5.根據權利要求4所述的基因組測序數據快速注釋方法,其特征在于,所述功能組件區包括啟動子區、外顯子區、內含子區、promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis、重復區和mirna區。6.一種基于劃窗映射的基因組測序數據快速注釋系統,其特征在于,包括以下模塊:索引庫模塊,用于存儲索引文件,其中,所述索引文件的構建方法如下:獲得測序樣本來源物種的功能組件區的起始位點和終止位點,對于每一個功能組件,利用公式(1)獲得映射值:其中,g
i
代表第i個位點的映射值,int表示取整數,s
i
代表第i個位點數值,s
i
代表第i個
位點所屬的劃窗區間的起始位點,w代表劃窗大小,w大小根據所述來源物種的所有功能組件區長度確定,由此獲得所有功能組件區的起始位點和終止位點的映射值以及位于功能組件區內的窗口起點/終點的映射值,按下列格式構建索引文件:chr s e s e function其中,chr代表功能組件區所在染體位置信息,s代表功能組件區起始位點或者位于功能組件區內的劃窗起點的映射值,e代表功能組件區終止位點或者位于功能組件區內的劃窗終點的映射值,s代表功能組件區起始位點,e代表功能組件區終止位點,function代表功能組件區的類別,輸入模塊,用于接收測序數據,獲得待注釋位點,并利用公式(1)計算待注釋位點的索引值,搜索模塊,分別與輸入模塊和索引庫模塊連接,用于將輸入模塊獲得的所述待注釋位點的索引值在所述索引文件的第2列和第3列進行搜索,若對于某一行j,g滿足s
j
≤g≤e
j
,進一步判斷q是否滿足s
j
≤q≤e
j
,若滿足,則所述待注釋位點可注釋位于第j行對應的功能組件區,結果輸出模塊,用于將注釋結果輸出。7.根據權利要求6所述的基因組測序數據快速注釋系統,其特征在于,所述w的確定方法具體如下:(1)獲得所述來源物種的所有功能組件區的長度;(2)獲得所有功能組件區長度的代表值,根據代表值確定w值。8.根據權利要求7所述的基因組測序數據快速注釋系統,其特征在于,所述代表值選自中位數、眾數、平均數、1/4分位數和3/4分位數中的一種;或者所述w值位于所述1/4分位數和3/4分位數之間。9.根據權利要求6所述的基因組測序數據快速注釋系統,其特征在于,所述來源物種為哺乳動物。10.根據權利要求9所述的基因組測序數據快速注釋系統,其特征在于,所述功能組件區包括啟動子區、外顯子區、內含子區、promoter cgis、intragenic cgis、3'transcript cgis、intergenic cgis、重復區和mirna區。
技術總結
本發明公開了一種基于劃窗映射的基因組測序數據快速注釋方法和系統,屬于生物信息技術領域。本發明首先將所有功能組件的起始位點和終止位點及位于功能組件中的劃窗起點和終點構建映射值,并利用該映射值建立索引文件,針對待注釋位點,同樣獲得映射值,進一步在索引文件中搜索映射值,若落在某一功能組件的對應的起點劃窗映射值和終點劃窗映射值中間,進一步判斷所有待注釋位點是否落在該功能組件的起始位點和終止位點之間,從而進行注釋。利用本發明,可以大大提高搜索注釋的效率,降低注釋的時間成本和計算成本。注釋的時間成本和計算成本。注釋的時間成本和計算成本。